Pythonでtweetを収集する
~~~追記~~~
2021/12/18 6:46
エラーが出て動かない
久々に使ったのでいつからエラーが出るようになったか不明
twint.token.RefreshTokenException: Could not find the Guest token in HTML
原因を調べてわかったら記事を書き直します。
~~~追記ここまで~~~
~~~追記その2~~~
2022/05/19 8:12
twintの開発が止まっているようです
self-development.info
別の方法を考えます
~~~追記その2ここまで~~~
twintを使う
twint · PyPI
インストール
$ pip3 install twint
twintはコマンドライン、pythonスクリプトの両方から使える
例えば@usernameのツイートから
Donald Trunpに関係するツイートを収集して
json形式のファイルに保存する
コマンドラインからは以下のようにする
$ twint -u username -s "Donald Trump" -o file.json --json
import twint, json path_json = 'c:\\path\\to\\json\\file.json' with open(path_json, 'w'): pass c = twint.Config() c.Username = "username" c.Search = "Donald Trump" c.Hide_output = True c.Store_json = True c.Output = path_json twint.run.Search(c) json_dict = [] with open(path_json, 'r', encoding = 'utf-8') as f: for line in f: json_dict.append(json.loads(line))
あとは出力されたjsonファイルを
煮るなり焼くなり好きに処理する
実行時に
>
CRITICAL:root:twint.get:User:'NoneType' object is not subscriptable + Unicode<
といったエラーが出る場合は
twintをインストールし直す
CRITICAL:root:twint.get:User:'NoneType' object is not subscriptable + Unicode issues · Issue #384 · twintproject/twint · GitHub
pip3 install --upgrade -e git+https://github.com/twintproject/twint.git@origin/master#egg=twint
あと、Jsonファイルはcp932でエンコードされているが
pythonで読み込むとエラーになる
それでencodingにutf-8を指定している
windowsだとこういう方法もある
【Windows】PythonでCP932(Shift-JIS)エンコード以外のファイルを開くとエラーになる問題がとりあえずの解決に至った件 - Qiita
Rでboxplotとbeeswarmを重ねて表示、その2
Rを使ってboxplotとbeeswarmを重ねて表示する
一度記事を書いているので今回はその2
mecobalamin.hatenablog.com
時系列データを曜日でまとめてbeeswarmでプロットする
こんなグラフができる
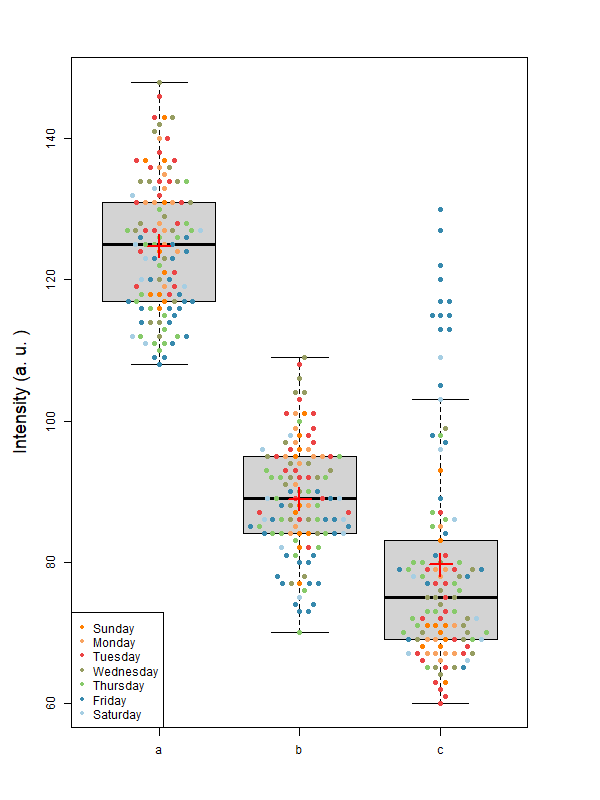
元のデータはこれと同じフォーマット
mecobalamin.hatenablog.com
点の数が増えている
boxplotとbeeswarmもpythonで書きたかったが
重ねて書く方法がわからなかったので
まずはRで描いてみる
ポイントは
- Deteから曜日を抽出する
- 曜日ごとにカラーコードを割り当てる
コードの流れはこんな感じ
- Dateをstrからdateに変換する
- 曜日の抽出
- 曜日にカラーパレットを割り当てる
- 曜日の数字を単語に置き換える
1. Dateをstrからdateに変換する
時系列のデータは1列目に日付が入っている
読み込んだときはstrなのでdateに変換する
この時、時間の起点を指定する
d$Date <- as.Date(d$Date, origin="1899-12-30")
1899なのは理由があるらしい
エクセルの日付(シリアル値)を、Rで使えるように変換する - Rプログラミングの小ネタ
エクセルの日時変数をRで扱う - Qiita
How to determine the correct argument for origin in as.Date, R - Stack Overflow
2. 曜日の抽出
as.POSIXltを使って曜日を抽出する
d$weekday <- as.POSIXlt(d$Date)$wday + 1
曜日は数字として得られる
3. 曜日にカラーパレットを割り当てる
8色つかってからパレットを作成して
そこから7色のカラーコードを取り出す
brewer.palで指定できる色数はdisplay.brewer.all()で表示されるパレットの色数で決まる
Pairedの場合は12まで指定できる
func_color_palette <- colorRampPalette(rev(RColorBrewer::brewer.pal(8, "Paired"))) color_palette <- func_color_palette(7)
曜日の数字を使って曜日にカラーコードを割り当てる
データごとに対応するカラーコードの列を作る
今回は3つデータの列があるのでカラーコードも3列作る
d <- d[order(d$weekday), ] d$color1 <- color_palette[d$weekday] d$color2 <- d$color1 d$color3 <- d$color1
4. 曜日の数字を単語に置き換える
weekdayの列を数字から単語に置き換える
ぐぐったらいくつかのサイトで似たような方法が出てきた
例えばこちらのサイト。
https://www.it-swarm-ja.tech/ja/r/%E6%9B%9C%E6%97%A5%E3%82%92%E6%8E%A2%E3%81%99/942412834/
この書き方をしたことなかったが
シンプルな書き方でいい感じ
d$weekday <- c("Sunday", "Monday", "Tuesday", "Wednesday", "Thursday", "Friday", "Saturday")[d$weekday]
グラフを書くだけならRが楽かな
pythonに置き換えられたらいいのだが。
ライブラリの読み込みを明示していないが
以下の2行ではライブラリを使っている
RColorBrewerとbeeswarmのインストールが必要
RColorBrewer::brewer.pal() beeswarm::beeswarm()
コードの全文
options(encoding = "UTF-8") rm(list = ls(all = True)) setwd("C:\\path\\to\\textfile") options(digits = 4, width = 100) bp_file_name <- "test.txt" func_color_palette <- colorRampPalette(rev(RColorBrewer::brewer.pal(8, "Paired"))) color_palette <- func_color_palette(7) d <- read.table(test, header = T, sep=",") d$Date <- as.Date(d$Date, origin="1899-12-30") d$weekday <- as.POSIXlt(d$Date)$wday + 1 d <- d[order(d$weekday), ] d$color1 <- color_palette[d$weekday] d$color2 <- d$color1 d$color3 <- d$color1 d$weekday <- c("Sunday", "Monday", "Tuesday", "Wednesday", "Thursday", "Friday", "Saturday")[d$weekday] pngname = sprintf("boxplot.png") png(pngname, width = 600, height = 800) par(mar = c(5, 5, 4, 5)) boxplot(d[2:4], ylab = "intensity (a. u.)", cex.lab = 1.5, outline = FALSE, methods = "hex" ) beeswarm::beeswarm(d[2:4], pwcol = d[7:9], add = TRUE, cex = 1.5, pch = 20) points(apply(d[,2:4], 2, mean), col = "red", pch = 3, lwd = 2, cex = 3) legend("bottomleft", legend = unique(d$weekday), pch = rep(20, length(unique(d$weekday))), col = unique(d$color1) ) dev.off() warnings()
*** 23 October 2021追記 ***
存在するオブジェクトを削除するコマンドを2行目で実行している
rm(list = ls(all =TRUE))
これは
R初心者のためのABC | A.ジュール, E.イエノウ, E.ミースターズ |本 | 通販 | Amazon
のp. 25に記述あり
*** 追記ここまで ***
メモリの換装
メモリを換装した
オリジナルより動作クロックの高いメモリが無事動いたのでメモ
PCはAsusのノートPCのUX310UAK
スペックはこんな感じ
| CPU | 第7世代 インテル Core i5 7200U(Kaby Lake) 2.5GHz/2コア | |
| 画面サイズ | 13.3 インチ | |
| メモリ容量 | 8GB | |
| メモリ規格 | DDR4 PC4-17000 |
ストレージは交換済み
もともとHDDとSSDが1台ずつだったが
SSD2台に交換してある
HDD: 500GB(SATA) -> SSD: 960GB(SATA)
SSD: 256GB(SATA) -> SSD: 1TB(NVMe)
オンボードで8GBのメモリが乗ってて
それとは別にメモリスロットが一つ空いていたので
16GBのメモリを刺してある
Timetec 16GB DDR4-2133 SODIMM
www.timetecinc.com
購入時にこの機種はHDDの交換やメモリの増設が
できることを調べてあった
分解動画も割と見つかる
分解に使うドライバーも購入済み

アネックス(ANEX) スーパーフィット 精密ヘクスローブドライバー T5×30 No.3542
- メディア: Tools & Hardware
1年ほど使ってて特に不満もなかったけれど
メモリも安くなってきたし
AmazonのBlack Fridayでポイントもつくしということで
16GBのメモリを32GBのメモリに交換した
問題は動作クロックが異なること
SO.DIMM DDR4 32GBのメモリで
2133MHz駆動のメモリが売ってない
調べた感じだと低い方のクロックに合わせて
動いてくれるそうなので
試しにTrasendの2666MHzで動くメモリを買って取り付けた
結果はWindowsが起動して40GB認識している


CPU-Zの見方がわからないので
正しい動作かどうかわからないが
今のところBSoDは見てない
このスペックの変化以外体感で
変わることもなく趣味の世界
画像処理なんかさせるとなにか体感がかわるかな
pythonで時系列の線グラフを作る

Pythonを使って上のようなグラフを作る
前回はヒストグラムだったが今回は線グラフ
mecobalamin.hatenablog.com
元のデータは以下のようなCSV形式
Date, a, b, c, d 2020/11/24 16:42:20, 143, 101, 63, 2020/11/24 17:27:19, 137, 101, 65, 2020/11/25 06:31:48, 134, 90, 65, 2020/11/25 11:21:57, 142, 109, 67, 2020/11/25 14:06:42, 131, 94, 74, 2020/11/25 18:54:18, 120, 91, 69, 2020/11/25 20:42:53, 114, 78, 72, 2020/11/26 09:09:53, 113, 84, 76, 2020/11/26 09:17:28, 125, 70, 70, 1021.67 2020/11/26 16:12:06, 126, 89, 65, 1019.64 2020/11/27 06:37:47, 117, 81, 69, 1020.66 2020/11/27 13:31:37, 123, 88, 98, 1019.98 2020/11/27 14:22:03, 126, 86, 89, 1019.64 2020/11/27 17:58:24, 120, 86, 84, 1020.32 2020/11/27 19:35:42, 116, 81, 81, 1021
1行目はヘッダーで
2行目以降がデータになっている
データの型は
1列目に日付と時間、2-4列目は整数、
5列目は浮動小数点数である
スクリプトの流れはこのようにした
また、ファイルを読み込むクラスと
グラフを作成するクラスを定義した
クラスの作成にはこちらの記事を参考にした
Pythonのオブジェクト指向プログラミングを完全理解 - Qiita
1. 定数の定義
まず入力するCSVファイル名やディレクトリを定義する
出力する画像ファイルに日付を付けるため、
日付から数字の文字列を作成する
date_today = datetime.date.today() day_save_file = str(date_today.year) + str(date_today.month).zfill(2) + str(date_today.day).zfill(2)
zfill()は0埋めをするメソッド
2. 出力するディレクトリの作成
ReadFiles().get_current_path()
スクリプトのあるディレクトリに
グラフを保存するディレクトリを作成する
os.getcwd()、os.chdir()でスクリプトのあるディレクトリを
作業ディレクトリに変更する
ReadFiles().create_directories()
作業ディレクトリにグラフ画像を保存するディレクトリを作成するが
try文を使ってディレクトリの有無を確認している
3. CSVファイルの読み込み
ReadFiles().read_files()
CSVファイルの読み込みにも
try文を使ってファイルが開けなかった場合の
エラーコードを表示させている
ファイルの読み込みには
pandasのread_csv()を使って
CSVをDataFrameとして読み込む
4. 1列目の型変換
読み込んだデータの1列目は日付と時間だが
データの型は文字列になっている
print(type(df['Date'][1])) <class 'str'>
to_datetime()を使って型を変換する
df['Date'] = pd.to_datetime(df['Date'])
変換するとTimestampになる
print(type(df['Date'][1])) <class 'pandas._libs.tslibs.timestamps.Timestamp'>
5. グラフの作成
読み込んだDataFrameからグラフを作る
CreateGraphs(df).create_line_graph()
まだ理解がぼんやりしているが
CreateGraphs(df)でインスタンスを作成し、
create_line_graph()のメソッドを呼び出している
ということになるらしい
create_line_graph()でグラフを描画する
twinx()で左右に軸を描いている
plt.ylim()が2度で使われている
最初のが左のy軸で次のが右のy軸になる
plt.ylim()の次に実行する
ax1.plot()及びax2.plot()のylim()を変更している
同じコマンドなので理解に時間がかかった
plot()のあとに軸の書き換えをするのに慣れない
パラメータを決めて一度に図を描画するのではなく
コマンドを追加して次々に書き換えていく
なのでplot()コマンドを最後にするものだと
思い込んでいたがそうでもなかった
描画したグラフはpngファイルとして保存される
plt.savefig(path_output_dir + '\\' + output_graph_name)
スクリプト全文は以下のようになる
元のデータをpython_sample.csvとして
スクリプトと同じディレクトリに保存し
スクリプトを実効するとグラフを作れる
# -*- coding: utf-8 -*- import os, sys, datetime from pandas import Series, DataFrame import pandas as pd import matplotlib.pyplot as plt import matplotlib.dates as mdates class ReadFiles: def __init__(self): pass def get_current_path(self): current_path = os.getcwd() current_path = os.chdir(os.path.dirname(os.path.abspath(__file__))) current_path = os.getcwd() return(current_path) def create_directories(self, path_to_dir): try: os.makedirs(path_to_dir) except FileExistsError: pass def read_files(self, path_to_file): print('open a csv file.') try: df = pd.read_csv(path_to_file) except Exception as e: print('cannot open file: ', path_to_file) print(str(e)) sys.exit(1) else: print('Success!') return(df) class CreateGraphs: def __init__(self, graph_data): self.graph_data = graph_data def create_line_graph(self): print('Create line plot') df = self.graph_data fig, ax1 = plt.subplots() plt.subplots_adjust(left=0.125, right=0.9, bottom=0.3, top=0.9) plt.ylim(50, 200) ax1.plot(df.index, df.iloc[:, 0], linestyle = '-', marker = 'o', color = 'r', label = df.columns[0]) ax1.plot(df.index, df.iloc[:, 1], linestyle = ':', marker = '.', color = 'b', label = df.columns[1]) ax1.plot(df.index, df.iloc[:, 2], linestyle = '--', marker = 's', color = 'y', label = df.columns[2]) ax2 = ax1.twinx() plt.ylim(1000, 1030) ax2.plot(df.index, df.iloc[:, 3], linestyle = ':', marker = '+', color = 'gray', label = df.columns[3]) locator = mdates.AutoDateLocator() formatter = mdates.DateFormatter('%m/%d') ax1.set_xticklabels(df.index, rotation = 90) ax1.xaxis.set_major_locator(locator) ax1.xaxis.set_major_formatter(formatter) handles1, labels1 = ax1.get_legend_handles_labels() handles2, labels2 = ax2.get_legend_handles_labels() ax1.legend(handles1 + handles2, labels1 + labels2, loc = 'upper left') if __name__ == '__main__': date_today = datetime.date.today() day_save_file = str(date_today.year) + str(date_today.month).zfill(2) + str(date_today.day).zfill(2) input_file_name = 'python_sample.csv' output_dir_name = 'graph' output_graph_name = day_save_file + '_graph_sample.png' path_input_file = ReadFiles().get_current_path() + '\\' + input_file_name path_output_dir = ReadFiles().get_current_path() + '\\' + output_dir_name + '\\' ReadFiles().create_directories(path_output_dir) df = ReadFiles().read_files(path_input_file) df['Date'] = pd.to_datetime(df['Date']) df = df.set_index('Date') CreateGraphs(df).create_line_graph() plt.savefig(path_output_dir + '\\' + output_graph_name) plt.close() print('Operation completed')
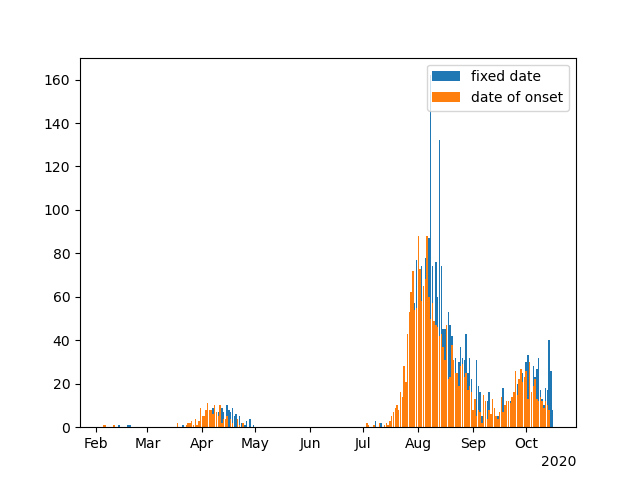
メモ:Pythonで時系列グラフの作成
時系列のデータをグラフにするとき
ハマったところをメモ
参考にしたサイト
note.nkmk.me
oku.edu.mie-u.ac.jp
元のデータはこれを使った
陽性者一覧(CSV:175KB)
csvのカラムは以下のようになっている
| 確定陽性者 | 性別 | 年齢 | 発病日 | 確定日 | 居住地 | 職業 | 推定感染経路 |
確定日に日付が入っているのでその数をカウントすると1日あたりの陽性者数を数えられる
それはpythonのvalue_counts()を使えばいいのだが
陽性者のいない日付もあるのでそこはNaNで埋めて日付が飛ばないようにしたい
初めは日付だけのデータフレームを作ってマージしたらよいかと思ったがうまくいかない
日付のデータフレームはDataFrame(pandas.date_range('日付1', '日付2'))を使うと
日付1から日付2の範囲の作れる
例えば次のスクリプトを実行する
from pandas import Series, DataFrame import pandas as pd import numpy as np date1 = DataFrame(pd.date_range('2020/2/1', '2020/2/7')) tmp_df = [] for i in date1.index: tmp_df.append('date1') tmp_df = DataFrame(tmp_df) date1 = pd.concat([date1, tmp_df], axis = 1) date1.columns = ['date', 'values'] date1.set_index('date', inplace = True) date1.drop(date1.index[[4, 5]], inplace = True) print(date1)
出力では5日と6日が抜けている
values date 2020-02-01 date1 2020-02-02 date1 2020-02-03 date1 2020-02-04 date1 2020-02-07 date1
更にインデックスを書き換えると
new_idx = pd.date_range(date1.index[0], date1.index[-1], freq = 'D') print(type(new_idx[0])) print(date1.index) date1 = date1.asfreq('D') print(date1)
なかった5日と6日の行にはNaNが挿入されている
values date 2020-02-01 date1 2020-02-02 date1 2020-02-03 date1 2020-02-04 date1 2020-02-05 NaN 2020-02-06 NaN 2020-02-07 date1
更に日付の範囲の異なるデータを作る
date1とdate2をpandas.concat()をつかって列で合成する
date2 = DataFrame(pd.date_range('2020/2/3', '2020/2/8')) tmp_df = [] for i in date2.index: tmp_df.append('date2') tmp_df = DataFrame(tmp_df) date2 = pd.concat([date2, tmp_df], axis = 1) date2.columns = ['date', 'values'] date2.set_index('date', inplace = True) print(date2) date = pd.concat([date1, date2], axis = 1) print(date)
結果は次の通り
values values date 2020-02-01 date1 NaN 2020-02-02 date1 NaN 2020-02-03 date1 date2 2020-02-04 date1 date2 2020-02-05 NaN date2 2020-02-06 NaN date2 2020-02-07 date1 date2 2020-02-08 NaN date2
date1とdate2それぞれにNaNの補完があって合成されている
同じようにしてvalue_counts()で日付の頻度をカウントして
日付をindexにしたらいいかと思ったが
このindexの日付とdate_range()で作った日付をconcat()で合成できない
できないというか日付の取り扱いが違っているようでうまくできない
ここまで書いてなんだが別の方法を試した
時系列データの場合、asfreq()でリサンプリングすることが可能
時系列の入ったデータフレームdf_dateを以下のようにすると
日付でデータを補完する
df_date = df_date.asfreq('D')
引数によってオフセットが変わるらしい
pandas.DataFrame.asfreq — pandas 1.5.3 documentation
Time series / date functionality — pandas 1.5.3 documentation
これを使って時系列のデータセットを作り
グラフを作るスクリプトが以下の通り
# -*- coding: utf-8 -*- import os, sys, datetime from pandas import Series, DataFrame import pandas as pd import numpy as np import matplotlib as mpl import matplotlib.pyplot as plt import matplotlib.dates as mdates def Count_Numbers(df_date, col_name): df_date = df_date.replace({'確認中': np.nan, '無症状': np.nan, '調査中': np.nan}) df_date = df_date.dropna() for n, i in enumerate(df_date): df_date[n] = '2020年' + df_date[n] df_date = pd.to_datetime(df_date, format = '%Y年%m月%d日') df_date = df_date.value_counts().sort_index() df_date = DataFrame(df_date) df_date = df_date.asfreq('D') df_date.columns = [col_name] return df_date if __name__ == '__main__': path = os.getcwd() path = os.chdir(os.path.dirname(os.path.abspath(__file__))) path = os.getcwd() date_today = datetime.date.today() day_save_file = str(date_today.year) + str(date_today.month) + str(date_today.day) arg = path + '\\' + 'Positives_List\\youseisyaitiran.csv' try: df = pd.read_csv(arg, index_col = 0, encoding = 'shift_jis') except Exception as e: print('cannot open file: ', arg) print(str(e)) sys.exit(1) else: print('we were able to open the file') fixed_date = Count_Numbers(df.iloc[:, 3], 'fixed_date') onset_date = Count_Numbers(df.iloc[:, 2], 'onset_date') tmp_date = pd.concat([fixed_date, onset_date], axis = 1) print(tmp_date) fig, ax = plt.subplots() locator = mdates.AutoDateLocator() formatter = mdates.ConciseDateFormatter(locator) ax.xaxis.set_major_locator(locator) ax.xaxis.set_major_formatter(formatter) ax.bar(tmp_date.index, tmp_date['fixed_date']) ax.bar(tmp_date.index, tmp_date['onset_date']) ax.legend(['fixed date', 'date of onset']) #ax.set_xticks() plt.savefig(path + '\\' + day_save_file + '_graph_bar.png') plt.close()
日付のないセルはNaNに置き換えている
グラフを作る命令はまだよくわかっていないが
このようにするといい感じのグラフができる
JBrowseをDockerで動かす、その7、イメージの作成
mecobalamin.hatenablog.com
続きです
前回までに作ったコンテナをイメージにする
イメージにするとインストールしたコマンドなどが保存される
使用しているコンテナは以下のコマンドで起動しているとする
$ docker run --rm -v /e/hoge:/tmp/fuga -v JBrowse:/usr/share/nginx/html/ -it --name jbrowse jbrowse
まず動いているコンテナの確認
wslから次のコマンドを実行する
以下wslから実行する
$ docker ps -a
コンテナの名前は起動時のnameオプションで付けた
jbrowseとなっているはず
jbrowseというイメージを作る
docker commitコマンドで
$ docker commit jbrowse jbrowse:1.0
jbrowseというコンテナに1.0というタグをつけて
jbrowseというイメージにした
確認は
$ docker images
でできる
ここまででイメージの作成ができた
イメージは使うイメージにタグを付けて起動できる
$ docker run --rm -v /e/hoge/:/tmp/fuga -v JBrowse:/usr/share/nginx/html/ -it -name jbrowse_new jbrowse:1.0
ここまででJBrowseをdockerのubutuにインストールして
ブラウザで確認する環境ができた
関連するコマンド
コンテナの終了
$ docker stop
docker runでrmオプションを付けていると
終了時にコンテナの削除もされる
イメージに残していない場合はインストールした
コマンド・ファイルは当然消える
コンテナの再開
$ docker start -i [コンテナ名]
iオプションをつけてインタラクティブモードで起動する
docker startコマンド - Qiita
コンテナの削除
$ docker rm [コンテナ名 or コンテナID]
docker runにrmオプションつけていないときに使う
イメージの削除
$ docker rmi [イメージID or リポジトリ:タグ]
JBrowseをDockerで動かす、その6、bamファイルの登録
mecobalamin.hatenablog.com
続きです
前回はJBrowseのテストデータを表示した
今回は自作のbamファイルをJBrowseに表示する
bamファイルは以下のように作った
mecobalamin.hatenablog.com
できたのは次の2ファイル
sort_SRR6799791.bam
sort_SRR6799791.bam.bai
比較につかうヒトゲノムのgff3ファイルと
faファイルは以下のように作った
mecobalamin.hatenablog.com
できたのは次の2ファイル
merge_chrom.fa
ref_GRCh38.p12_top_level.gff3
この4ファイルを使う
まずmerge_chrom.faとref_GRCh38.p12_top_level.gff3を
ホストOSからJBrowseのボリュームにコピーする
コピーのやり方だが例えば以下の記事ではホストOSと
ボリュームの両方にマウントしつつ
dockerのUbuntuを起動しているのでコピーできる
JBrowseをDockerで動かす、その4、JBrowseのインストール - mecobalamin’s diary
faとgff3を保存するディレクトリは何でもいいがここではdbとする
JBrowseをインストールしたディレクトリに移動し
以下のようにスクリプトを実行して
faとgff3ファイルを登録する
$ cd /var/share/nginx/html/jbrowse $ bin/prepare-refseqs.pl --fasta ../db/merge_chrom.fa --out HeLa_SRR6799791_dev --trackLabel GRCh38_latest_genomic --seqType dna $ bin/flatfile-to-json.pl --gff ../db/ref_GRCh38.p12_top_level.gff3 --trackType CanvasFeatures --out HeLa_SRR6799791_dev --trackLabel ref_GRCh38
次にbamファイルの登録する
上のスクリプトを実行すると配列を保存するディレクトリが作られる
この場合はHeLa_SRR6799791_devである
このディレクトリにbamというディレクトリを作り
手持ちのbamとbam.baiをコピーする
そして以下のスクリプトを実行する
$ cd HeLa_SRR6799791_dev $ ../bin/add-bam-track.pl --label HeLa_SRR6799791 --bam_url bam/sort_SRR6799791.bam --in trackList.json
ホストOSのブラウザから以下のアドレスにアクセスすると
登録したbamファイルを見られる
http://192.168.0.4:8000/jbrowse/index.html?data=HeLa_SRR6799791_dev
192.168.0.4はホストのIPアドレスで
ホストのIPアドレスはwslではifconfigを使う
とりあえずここまでで手持ちのbamファイルを表示できた
