apt-getでファイルがダウンロードできない
ライブラリを追加したくてapt-getを使ったら
ファイルがダウンロードできずにエラーが出たので
解決方法のメモしとく
欲しかったのはlibGL.so.1
opencv_createsamplesをインストールして実行したら
libGL.so.1がないってエラーが出た
パッケージの探し方もよくわからず。。。
ググるとパッケージ検索のサイトが見つかった
Ubuntu – Ubuntu Packages Search
"パッケージの内容を検索"
でキーワードに
"libGL.so.1"
を入れて
"キーワードに似た名前のファイルが含まれるパッケージ"
を探す
Ubuntu – Package Contents Search Results -- libGL.so.1
libgl1で良さそうなのでapt-getでインストール
$ apt-get install libgl1
だけど404エラーがでて必要なファイルがダウンロードできず。。。
解決方法がよくわからなかったけど、
apt-getのupdateで良かったらしい
$ sudo apt-get update
update後にapt-getを実行
$ sudo apt-get install libgl1
今回はエラーも出ず、
無事インストールできた
ググると出てきたのはインストール元を指定するファイルが
古くてダウンロードできないときの解決方法が多いみたいだった
apt-get updateが404で動かない場合もあるらしい
https://server.etutsplus.com/apt-get-404-not-found/
メモ: リードカウントデータの正規化について
17 May 2019追記
メモした内容で実際に正規化してみた
mecobalamin.hatenablog.com
mecobalamin.hatenablog.com
追記ここまで
featureCountsでリードカウントしたあとに
複数のデータを比較する場合、
カウントの正規化が必要らしい
まだ良くわかってないので
参考にしているサイトのリンクを張っておく
ペアエンドのサンプルをリードカウントするときのコマンドがあった
featureCounts
RNA seqのリードカウント featureCounts - macでインフォマティクス
正規化の方法にはRLE正規化とTMM正規化があって、
求める正規化のファクターが異なる
これらの正規化方法を利用してるのがDESeq/DESeq2やedgeRで、
Rのライブラリーとして配布されている
DESeq/DESeq2はRLE正規化を使う
RLE正規化
RLE 正規化 | DESeq/DESeq2 に実装されている RNA-Seq カウントデータの正規化法
edgeRはTMM正規化を使う
TMM正規化
TMM 正規化 | edgeR で発現変動遺伝子を検出する際に利用する RNA-Seq データの正規化法
DESeq2を使った正規化でサンプルの処理をしている
サルマップ2018 (3) DESeq2による標準化と可視化まで
サルマップ2018 (3) DESeq2による標準化と可視化まで - ノンコーディングRNAネオタクソノミ
こっちはedgeR
edgeR - macでインフォマティクス
28 April 2019追記
edgeR: リードカウントから発現変動遺伝子を検出 - Heavy Watal
こちらにあったがedgeRのUser's Guideが詳しい
まだ読んでる途中だがCase Studiesもある
library(edgeR) edgeRUsersGuide(view=TRUE)
でpdfを表示できる
なおedgeR、DESeq2ともにアップデートがあったようだ
それについてはこの記事の最後に追記した
追記ここまで
edgeRとDESeq2はbioconductorを使ってインストールする
Bioconductor - Home
パッケージマネージャーのBiocManagerをインストールしてから
ライブラリをインストールする
RはwindowsのRGuiを使っていて
versionは3.5.2
BiocManagerのインストール
Bioconductor - Install
edgeRのインストール
Bioconductor - edgeR
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("edgeR", version = "3.8")
DESeq2のインストール
Bioconductor - DESeq2
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("DESeq2", version = "3.8")
とりあえずライブラリのインストールまで済んだ
28 April 2019追記、その2
edgeR、DESeq2ともにアップデートがあったようだ
インストールのコマンド
if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager") BiocManager::install("edgeR", version = "3.8")
を再実行するといくつかのパッケージがアップデートされた
それに伴いRのパッケージも手動でアップデートする必要があった
例えば
library(edgeR)
でlibraryを読み込ませたときに
足りないパッケージが表示される
(Rccpだったかな。。。)
RGuiの場合はツールバーから
[パッケージ]->[パッケージのインストール]を選択
先にCRANのミラーサイトを選択する
次にパッケージを選んでインストールする
コマンドラインからだと
install.packages("Rccp", dependencies = TRUE)
でインストールできる
このときCRANのミラーサイトが選ばれていなければ
選択するウィンドウが表示される
追記ここまで
hg38のfastaとgtf/gff3
GRCh38とhg38はヒトゲノムのシークエンスデータ
ほとんど一緒らしい。
前にも引用させてもらったけどここに説明がある。
GRCh38とhg38の違い(含むミトコンドリア)
GRCh38染色体名(ヘッダ行)が長く(gi|568815597|ref|NC_000001.11|)、hg38は簡潔(chr1)。
ヘッダ行に違いがあるのはやや問題で、
JBrowseで表示させたりfeatureCountsでリードカウントするときに
スムーズにできないことがあった
そこで簡潔な表示で揃えてfastaとgtf/gff3を作成する
まずはシークエンスデータのダウンロード
UCSC Genome Browser Downloads
リンクをたどるといくつかファイルが見つかる
chromosomeごとに分かれているデータを使う
Index of /goldenPath/hg38/bigZips
ファイルの説明はこちら
hg38.chromFa.tar.gz - The assembly sequence in one file per chromosome.
Repeats from RepeatMasker and Tandem Repeats Finder (with period
of 12 or less) are shown in lower case; non-repeating sequence is
shown in upper case.
ダウンロードと展開
$ wget http://hgdownload.soe.ucsc.edu/goldenPath/hg38/bigZips/hg38.chromFa.tar.gz $ gzip -d hg38.chromFa.tar.gz $ tar -xvf hg38.chromFa.tar
展開するとパッチを含めて450ファイルぐらいある
パッチは取り扱いがよくわからないので
とりあえずchromosomeのファイルだけマージして使う
$ :> merge_chrom.fa $ for i in {1..22}; do cat chr${i}.fa >> merge_chrom.fa; done $ cat chrX.fa >> merge_chrom.fa $ cat chrY.fa >> merge_chrom.fa
このfastaファイルをhisat2-build及びhisat2に使う
mecobalamin.hatenablog.com
次にhg38のgtfファイルを作成する
Gene annotation データを用意する(gtf形式) - Palmsonntagmorgen
NCBIからダウンロードできるgffファイルは詳しい表記のヘッダなので、
UCSCのサイトからgtfファイルをダウンロードしてgff3に変換する
Table Browser@UCSC
Table Browser
設定はこうした・・・ハズ。
clade: Mammal
genome: Human
assembry: Dec. 2013 (GRch38/hg38)
group: Gene and Gene Predictions
track: NCBI RefSeq
table: RefSeq All (ncbiRefSeq)
region: genome
output format: GTF - gene transfer format (limited)
output file: UCSC.hg38.gtf.gz
file type returned: gzip compressed
gtfファイルをgffreadをつかってgff3に変換する
$ gffread -E UCSC.hg38.gtf -o- > UCSC.hg38.gff3
このファイルはパッチで修正?された配列を含む
つまりパッチとchromosomeの間に重複がある
正しいやり方かよくわからないがパッチ側の重複した配列を除く
取り除かないと通らないコマンドがあった
bam2wigだったかな
$ cat UCSC.hg38.gff3 | awk '(a[$9]++ < 1000) && ($1 !~/[_alt]/) {print}' > rename_UCSC.hg38.gff3
exonやCDSで同じ名前のエントリーがあるので
1000個以上の重複で"_alt"を含む場合に除く
コードでは重複が1000個以下でかつ"_alt"を含まない行をファイルに出力している
1000個以上重複している配列はなさそうだったので、
使った感じでは取りこぼしはなさそう
今度はgff3をgtfに変換する
$ gffread rename_UCSC.hg38.gff3 -T -o- > rename_UCSC.hg38.gtf
gtfはfeatureCountsに、gff3はJBrowseに使う
ファイルの中身はこんな感じ
rename_UCSC.hg38.gtf
chr1 hg38_ncbiRefSeq exon 11874 12227 . + . transcript_id "NR_046018.2"; gene_id "NR_046018.2"; chr1 hg38_ncbiRefSeq exon 12613 12721 . + . transcript_id "NR_046018.2"; gene_id "NR_046018.2"; chr1 hg38_ncbiRefSeq exon 13221 14409 . + . transcript_id "NR_046018.2"; gene_id "NR_046018.2"; chr1 hg38_ncbiRefSeq exon 14362 14829 . - . transcript_id "NR_024540.1"; gene_id "NR_024540.1"; chr1 hg38_ncbiRefSeq exon 14970 15038 . - . transcript_id "NR_024540.1"; gene_id "NR_024540.1";
rename_UCSC.hg38.gff3
# gffread -E UCSC.hg38.gtf -o- # gffread v0.10.6 ##gff-version 3 chr1 hg38_ncbiRefSeq transcript 11874 14409 . + . ID=NR_046018.2;geneID=NR_046018.2 chr1 hg38_ncbiRefSeq exon 11874 12227 . + . Parent=NR_046018.2 chr1 hg38_ncbiRefSeq exon 12613 12721 . + . Parent=NR_046018.2 chr1 hg38_ncbiRefSeq exon 13221 14409 . + . Parent=NR_046018.2 chr1 hg38_ncbiRefSeq transcript 14362 29370 . - . ID=NR_024540.1;geneID=NR_024540.1 chr1 hg38_ncbiRefSeq exon 14362 14829 . - . Parent=NR_024540.1 chr1 hg38_ncbiRefSeq exon 14970 15038 . - . Parent=NR_024540.1
bigwigの作成と表示
JBrowseでbamを表示させるとリードがずらーっと並ぶ
実際にいくつあるのかわかりやすくするために
ヒストグラムで表示したい
bamをbigwig形式にする
bam2wigとwigToBigWigを使う
ここを参考にした
bamからbigWigとWiggle Formatに変換するツール - macでインフォマティクス
コマンドはここからダウンロード
bam2wig: bamからwigを作成
GitHub - MikeAxtell/bam2wig: Conversion of a BAM alignment to wiggle and bigwig coverage files, with flexible reporting options
wigToBigWig: wigからbigwigを作成
Index of /admin/exe/linux.x86_64
両方にパスを通す
wigToBigWigにパスが通っていたら
bam2wigは自動でwigToBigWigも実行する
変換はbam2wigにbamを読み込ませるだけ
$ bam2wig hogehoge.bam
こんな名前のディレクトリhogehoge_bam2wigが
作られてbigwigも出力される
できたbigwigファイルをjbrowseで表示するには
jbrowseのデータのあるディレクトリにコピーして
スクリプトで取り込ませる
How do I set up a BigWig file?
JBrowse FAQ - GMOD
$ add-bw-track.pl --label mybw --bam_url file.bw
Example BigWig-based Wiggle XY-Plot Track Configuration
JBrowse Configuration Guide - GMOD
例えば作ったファイルhogehoge.bigwigが
以下のディレクトリに存在する場合
$ /var/www/html/jbrowse/hogehoge/bw
コマンドを以下のように実行した
trackList.jsonのあるディレクトリに移動して
スクリプトを実行するのが良さそう
$ cd /var/www/html/jbrowse/hogehoge/ $ ../bin/add-bw-track.pl --label hogehoge_bigwig --bw_url bw/hogehoge.bigwig --in ./trackList.json
trackList.jsonをvimで編集
hogehoge.bigwigのあるエントリーで
typeの項目を書き換える
"type" : "JBrowse/View/Track/Wiggle/XYPlot"
登録した結果はこんな感じ
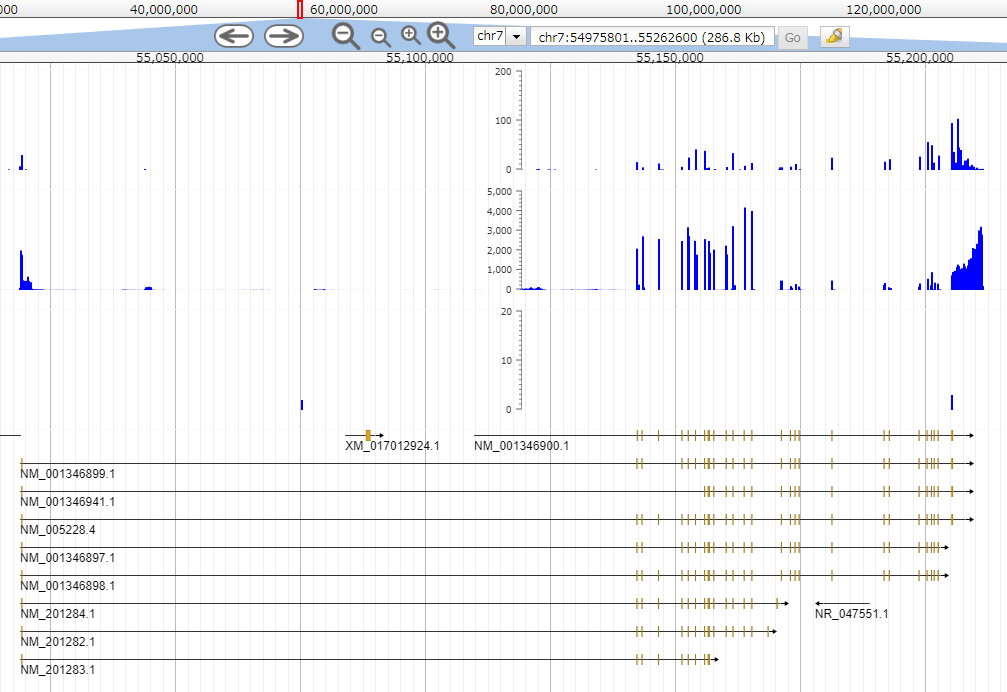
青のヒストグラムがbigwig
ヒストグラムの下の部分は
ヒトゲノムのgff3を表示している
ちなみに異なる3種類のbigwigファイルを表示しているので
ヒストグラムが3段ある
ヒトゲノムのfastaファイルとgffは
以前作ったファイルとは変わっている
前にもちょっと書いたけど公開されているヒトゲノムは
ヘッダーの書き方に違いがある
GRCh38とhg38の違い(含むミトコンドリア)
これはhisatで作ったbamファイルのラベルが変わってくる
あと、gtfファイルはUCSCのゲノムブラウザを使って作成する
Gene annotation データを用意する(gtf形式) - Palmsonntagmorgen
Table Browser
gtfファイルはgffreadを使ってgffに変換する
$ gffread -E hg38_ucsc.gtf -o- > hg38_ucsc.gff3
ちなみに逆もできる
$ gffread rename_UCSC.hg38.gff3 -T -o- > rename_UCSC.hg38.gtf
このgffファイルにはシークエンスデータのパッチも含まれる
これが意外に厄介でパッチで追加された配列に重複があると
いろいろ変換ができなくなる
問題ありそうだけど重複部分を削除することにした
良くないんだろうけど。。。
ゲノムデータのパッチの取扱がよくわからない。
重複部分の削除とJBrowseの登録はこんな感じ
$ cat UCSC.hg38.gff3 | awk '(a[$9]++ < 1000) && ($1 !~/[_alt]/) {print}' > rename_UCSC.hg38.gff3 $ bin/flatfile-to-json.pl --gff ~/usr/data/db/rename_UCSC.hg38.gff3 --trackType CanvasFeatures --out hogehoge --trackLabel ref_hg38_ch
詳しくはまたあとで。
20 April 2019追記
作り直したhg38のgtfファイルについてはここにまとめてある
mecobalamin.hatenablog.com
このファイルをJBrowseに登録したり
マッピングに使ったりした
追記ここまで
RNA-seqその6、featureCountsでリードカウント
前回作ったbamファイルを使って発現量を調べる
mecobalamin.hatenablog.com
使うコマンドはfeatureCount
こちらのサイトを参考にした
featureCount で bam と gtf からリードカウントを得る | Tips for NGS Data Analysis
https://ncrna.jp/blog/item/387-featurecount-2018
featureCountで検索かけるとRを使った解析ソフトウェア群の
Bioconductorが見つかる
Bioconductor - Home
BiocManagerというパッケージマネージャーもある。
(biocLiteはバージョンの古いマネージャー)
featureCountはRsubreadに含まれるが
こちらのwslでは動かなかった
なのでコマンドライン版のfeatureCountを使う
The Subread package
ここからダウンロードできる。
最新の1.6.3と、1.5.0-p2はこちらの環境では動かなかった
1.6.3は実行後エラーが出てすぐに止まった
1.5.0-p2はエラーメッセージが表示され続けた
1.5.3は動いたのでこのバージョンを使う
ファイルを展開後makeして$HOME/binにリンクを張った
WEHI Bioinformatics - Subread package
31 May 2019追記
最新版1.6.4が公開済みだった
アーカイブをダウンロードして展開
バイナリが含まれていたので$HOME/binにリンクを張った
ちゃんと動いた
追記ここまで
gtfファイルとbamファイルを読み込ませて実行する
結果はcounts.txtに出力される
$ featureCounts -p -O -T 4 -t exon -g gene_id -a ref_GRCh38.p12_top_level.gtf -o counts.txt hogehoge.bam
6 March 2019追記
- pと-Oオプションを追加
- pはペアエンド、-Oは重なりがあるリードがあるときに指定するらしい。
Assign reads to all their overlapping meta-features (or features if -f is specified).
featureCounts | 各遺伝子にマッピングされたリード数を計数
WEHI Bioinformatics - featureCounts
追記ここまで
このcounts.txtをRに読み込ませて解析するが、
とりあえずヒストグラムを作る
ここからはRで作業する
counts.txtのあるディレクトリに作業ディレクトリを移動し、
read.table()でファイルを読み込ませる
一行目のコメントとヘッダーを飛ばして、
タブ区切りにする
どれがカウントデータかよくわからないけど
colnames()で確認すると
[1] "Geneid" "Chr" "Start" "End" "Strand" [6] "Length" "hogehoge.bam"
なのでおそらく最後のカラム
試しにコピー数が3から30のだけを取り出して
ヒストグラムにする
setwd("/path/to/counts.txt") fc <- read.table("counts.txt", header = TRUE, skip = 1, sep="\t") d <- subset(fc, fc$hogehoge.bam < 30) d <- subset(d, d$hogehoge.bam > 3) h <- hist(d$hogehoge.bam)
結果はこんな感じ

ちなみにpng形式で保存するには
Rに表示されているグラフを消してから
png()のコマンドでファイル名とサイズを宣言
hist()を実行、そしてdev.off()でファイルを閉じる
png("histogram.png", width = 600, height = 450) h <- hist(d$hogehoge.bam, main = "", xlab= "hogehoge.bam") dev.off()
10 March 2019修正
dev.off()が抜けてたので追加
必要ならpng()の前にもdev.off()を実行して
null device
1が表示されてから上記のコマンドを実行する
hにはhistogramのパラメータが入っている
$breaksはhist()の引数breaksを指定することで
変更可能でbin幅が変えられる
> h $breaks [1] 4 6 8 10 12 14 16 18 20 22 24 26 28 30 $counts [1] 1114 358 250 196 154 134 87 104 93 64 54 51 34 $density [1] 0.206832529 0.066468622 0.046416636 0.036390642 0.028592648 0.024879317 0.016152989 [8] 0.019309320 0.017266988 0.011882659 0.010025993 0.009468994 0.006312662 $mids [1] 5 7 9 11 13 15 17 19 21 23 25 27 29 $xname [1] "d$hogehoge.bam" $equidist [1] TRUE attr(,"class") [1] "histogram"
次は目的の遺伝子がいくつあるか
グラフ化したい
11 March 2019追記
HeLaのbamファイルとhg38のgtfファイルを使ってfeatureCountsを試した
hg38のfastaとgtfはリンク先を参照
mecobalamin.hatenablog.com
HeLaのsraファイルはSRR6799791で、
hg38のシークエンスデータにhisat2を使ってマッピングした
できたファイルがHeLa.bam
featureCountsを実行する
$ featureCounts -p -B -T 4 -O -t exon -g gene_id -a rename_UCSC.hg38.gtf -o counts.txt HeLa.bam
できたcounts.txtから試しにEGFRの数を抜き出してみる
https://www.ncbi.nlm.nih.gov/gene/1956
上皮成長因子受容体 - Wikipedia
EGFRにはスプライシングバリアントがある
それぞれのaccession numberでカウントをcounts.txtから抜き出す
Rを使う
acc.egfr <- c("NM_005228", "NM_201282", "NM_201283", "NM_201284", "NM_001346897", "NM_001346898", "NM_001346899", "NM_001346900", "NM_001346941") iso.egfr <- c("a", "b", "c", "d", "e", "f", "g", "h", "i") var.egfr <- t(rbind(acc.egfr, iso.egfr)) fc <- read.table("counts.txt", header = TRUE, skip = 1, sep="\t") egfr <- c() for(i in var.egfr[,1]){egfr <- rbind(egfr, subset(fc, grepl(i, fc$Geneid)))} cbind(iso.egfr, egfr[c(1,7)])
結果はこうなったけど
これをそのままmRNAのコピー数としていいのか。。。?
iso.egfr Geneid HeLa.bam 134652 a NM_005228.4 981 134654 b NM_201282.1 254 134653 c NM_201283.1 190 134655 d NM_201284.1 255 134648 e NM_001346897.1 494 134649 f NM_001346898.1 494 134650 g NM_001346899.1 981 134657 h NM_001346900.1 963 134651 i NM_001346941.1 945
RNA-seqその5、Hisat2でマッピング
RNAseqの結果をリファレンス配列にマッピングする
mecobalamin.hatenablog.com
レファレンスデータはヒトのゲノムデータ
Human Genome Resources at NCBI - NCBI
ここからReference Genome Sequenceをダウンロード
28 Feb. 2019追記
ヒトゲノムデータにはデータの書き方の違いで2種類あるらしい
GRCh38とhg38の違い(含むミトコンドリア)
chromosomeの記述の仕方に違いがある
JBrowseで表示したり解析に使うにはhg38がいいのかも
Index of /goldenPath/hg38/bigZips
追記ここまで
20 April 2019追記
ヒトゲノムのデータを作り直した
mecobalamin.hatenablog.com
問題あるかもだけどchromosoneのファイルだけ一つにまとめて
インデックスファイルの作成に使用した
以下のスクリプトではname_dbとpostfixを書き換える
追記ここまで
まずヒトゲノムをhisat2-buildでインデックスファイルに変換する
#!/bin/bash echo "hisat2 build began" dir_scripts="$HOME/usr/scripts" dir_working="$HOME/usr/data" dir_db="${dir_working}/db" name_db="GRCh38_latest_genomic" postfix="fna" hisat2-build ${dir_db}/${name_db}.${postfix} ${name_db}
input file + 数字 + .ht2という形式で複数のファイルが出力される
GRCh38_latest_genomic.1.ht2
今回は8個だったけどいつも同じファイル数か不明
インデックスファイルができたらいよいよマッピング
hisat2の引数にインデックスファイルのパスと
ファイル名の数字の前までを指定する
マージしたFastqもリード1、2のオプションを付けて読み込ませる
samファイルで出力される
マッピングが終わるとsamtoolsでsamからbamにして
ソートしてindexをつける
stringtieでアノテーションを付ける、
とのことなんだけどよくわかってない
出力されたgtfファイルを見ると
bamに含まれる遺伝子名や場所をまとめたファイルのようだ
transcripだけ読めばカウントできそうだけどどうだろう
#!/bin/bash echo "hisat2 analysis" echo "set directories" dir_scripts="$HOME/usr/scripts" dir_working="$HOME/usr/data" dir_db="${dir_working}/db" name_db="GRCh38_latest_genomic" echo "open list sra" dir_list_sra=${dir_scripts} name_sra="list_sra.txt" list_filename=$(cat ${dir_list_sra}/${name_sra} | sed 's/\.sra//g') for i in ${list_filename} do echo "analyze ${i}" dir_out="hisat_${i}" mkdir -p ${dir_working}/${dir_out} echo "hisat began" hisat2 \ -p 4 \ -x ${dir_db}/${name_db} \ --rna-strandness RF \ --dta \ -1 ${dir_working}/trim_${i}/ps/ps_${i}_gd_1.fastq \ -2 ${dir_working}/trim_${i}/ps/ps_${i}_gd_2.fastq \ -S ${dir_working}/${dir_out}/hisat_${i}.sam \ echo "samtools began" samtools view -bS -@ 4 ${dir_working}/${dir_out}/hisat_${i}.sam |\ samtools fixmate -@ 4 - ${dir_working}/${dir_out}/fixmate_${i}.bam samtools sort ${dir_working}/${dir_out}/fixmate_${i}.bam \ -o ${dir_working}/${dir_out}/sort_${i}.bam \ -m 10G \ -@ 4 samtools index ${dir_working}/${dir_out}/sort_${i}.bam echo "stringtie began" stringtie \ ${dir_working}/${dir_out}/sort_${i}.bam \ -p 4 \ -o ${dir_working}/${dir_out}/stringtie_${i}.gtf \ -l stiringtie_${i} echo "analysis finished" done
こうやってできたbamファイルをJBrowseで表示した
18 April 2019追記
ファイルのサイズが大きい時にエラーが出てスクリプトが止まる
samtool sort の時とstringtieで起きた
samtool sortのメモリ指定を変えたほうがいいかも
ヘルプによると-m optionはmemory per threadとのこと
Windowsを起動して空きメモリは16〜20ぐらいなので
4Gとか5Gを指定するのがいいかも
試した感じでは止まらずに動いた
Stringtieは-cと-jオプションを使うといいらしい
github.com
どの程度の値がちょうど良いのか
生物学的な意味があるかもしれないが
とりあえず倍にした
-c 5 (default 2.5) -j 2 (default 1)
これでプログラム的にはオッケー
止まっていたファイルでも動くようになった
RNA-seqその4、Fastqファイルのマージ
クリーニングしたFastqファイルを一つにまとめる
mecobalamin.hatenablog.com
plinseq-liteで評価の良かったリードだけをマージする
ファイル名に"gd"を含むファイルを使うことになる
read 1とread 2を区別する
マージしたファイルは、マージ前と同じディレクトリに保存される
#!/bin/bash dir_scripts="$HOME/usr/scripts/" dir_working="$HOME/usr/data/" dir_list_sra=${dir_scripts} name_sra="list_sra.txt" list_filename=$(cat ${dir_list_sra}/${name_sra} | sed 's/\.sra//g') for i in ${list_filename} do dir_div_file="${dir_working}/raw/div_${i}" list_div_file="list_div_${i}.txt" list_read_1=$(cat ${dir_div_file}/${list_div_file} | grep _1_) list_read_2=$(cat ${dir_div_file}/${list_div_file} | grep _2_) echo "merge read 1" :> ${dir_working}/trim_${i}/ps/ps_${i}_gd_1.fastq for j in ${list_read_1} do cat ${dir_working}/trim_${i}/ps/ps_${j/_1_/_}_gd_1.fastq >> \ ${dir_working}/trim_${i}/ps/ps_${i}_gd_1.fastq #echo ${i}, ${j/_1_/_} done echo "merge read 2" :> ${dir_working}/trim_${i}/ps/ps_${i}_gd_2.fastq for j in ${list_read_2} do cat ${dir_working}/trim_${i}/ps/ps_${j/_2_/_}_gd_2.fastq >> \ ${dir_working}/trim_${i}/ps/ps_${i}_gd_2.fastq done done
14 April 2019追記
複数のファイルを処理していたら
trimmomaticのあとディスクの容量が足りなくなった
いくつか中間ファイルを消していたら
rawのディレクトリを間違って消してしまって
一緒に分割ファイルのリストも無くなった。。。
次に進めず。。。。
分割ファイルを再作成するスクリプト
mkdir $HOME/usr/data/raw/ f="$HOME/usr/scripts/list_sra.txt" fn=$(cat ${f} | sed 's/\.sra//g') for i in ${fn}; do mkdir $HOME/usr/data/raw/div_${i}; done for i in ${fn}; do for j in {1..2}; do for k in 01 02 03 04 05 06 07 08 09 10; do :> $HOME/usr/data/raw/div_${i}/div_${i}_${j}_${k}; done; done; done for i in ${fn}; do ls $HOME/usr/data/raw/div_${i}/ > $HOME/usr/data/raw/div_${i}/list_div_${i}.txt; done
空のファイル群を作成raw/に作成したあとに
ファイルのリストを作成する
あまりきれいな書き方ではないが一応大丈夫っぽい
コマンドライン上で動かしていたのでfor文も一行で書いている
このままシェルスクリプトファイルにしても動くかも
追記ここまで
20 April 2019追記
別の処理中にペアエンドのリードの数が1と2で違っている雰囲気があったので確認
前にも確認してたけど念のためもう一度
あとスクリプトの記録
$ cat hogehoge_gd_1.fastq | wc -l 61570060 $ cat hogehoge_gd_2.fastq | wc -l 61570060
リードの数は同じ
4の倍数かどうかも確認
割り算だと整数で返してくるので余りを求める
$ echo $((61570060%4)) 0
いくつかマージ後のファイルを調べたけど大丈夫そう
マージ前は?
$ for i in 01 02 03 04 05 06 07 08 09 10; do echo $(($(cat hogehoge_${i}_gd_1.fastq | wc -l)%4)); done
こっちも問題なさそう
追記ここまで