Atomでscript
普段使っているエディターはAtom
殆どいじっていないが、
pythonとRを実行できるようにしてある
必要なpackageは
scriptとlanguage-r
packageをインストールしたら
windowsのシステム環境変数で
Rscriptのディレクトリにパスを通す
\R-3.5.0\bin
windowsの設定 -> 環境変数を検索 -> システムのプロパティ -> 環境変数
または
コントロールパネル -> システム -> システムのプロパティ -> 環境変数
からpathを編集する
やってたはずだけど
windows10を再インストールしたときに
やり忘れてたみたいなので
これどっかに書いてあったはずだけど。。。
あとlanguage-rはそのままだとエラーが出たはず
ファイルの記入ミスだったと思う
修正方法を載せたサイト見つけたら追記予定
27 May 2019追記
まずcummulative maxでエラーが出る件についてはここを参考に修正する
Atom editor r-language error - Failed to load snippets - Stack Overflow
Duplicate key 'Cummulative max' · Issue #17 · REditorSupport/atom-language-r · GitHub
次にgrepのところでエラーが出るが、
同じキーが有るのが良くないっぽい
'Grep': 'prefix': 'grep' 'body': 'grep(${1:pattern}, ${2:x}, ${3:ignore.case = ${4:FALSE}}, ${5:perl = ${6:FALSE}})' 'Grep': 'prefix': 'grep' 'body': 'grep(${1:pattern}, ${2:x}, ${3:ignore.case = ${4:FALSE}}, ${5:perl = ${6:FALSE}}, ${7:value = ${8:FALSE}}, ${9:fixed = ${10:TRUE}})'
これでとりあえずエラーは出なくなった
追記ここまで
16 October 2020 追記
Atomの出力に日本語を表示する
言語ごとに設定が違うっぽいが
pythonの場合は
[file] -> [initial script] -> init.coffee]
に
process.env.PYTHONIOENCODING = "utf-8";
を追記する
追記ここまで
メモ: コマンドのリンク先とWSLのCPU使用率
condaでパッケージをインストールするときの注意点
pythonとパッケージをインストールしたはずなんだけど
コマンド実行中にモジュールが見つからないってエラーが出ることがある
実際Trinityを使っててnumpyがないってエラーがでた
パッケージを入れてあるか確認。
$ conda list
numpyの1.15.4がインストールされてる。
pythonは3.6.8
pythonを実行して確認するとバージョンが違う
$ python --version
Python 2.7.15rc1
/usr/binにインストールされてるpythonを使っていたようだ
ubuntuには予めpythonがインストールされてて
condaで入れたpython/packageとは別になる
さてどうしよう。。。
このときは~/bin以下にminiconda3で入れたpythonのリンクを貼った
~/.bashrcにこんな感じでpathを記入して~/binにpathを通す
すでに書かれている場合は追記で。
前ほど優先順位が高いので、他のプログラムで使っているコマンドとの兼ね合いもある
export PATH="$HOME/bin:/usr/bin:$HOME/miniconda3/bin:$PATH"
~/binにpythonリンクを貼る
$ ln -s $HOME/miniconda3/bin/python $HOME/bin
これでcondaで入れたpythonとパッケージを使えるようになる
それとWSLの問題について
WSLでコマンドを実行中にwindowsのタスクマネージャーを見ると
CPUの使用率がやたらと高い時がある
perlだったりubuntuだったりならわかるが
windows defender antivirus serviceの使用率が
30-40%程度になることがある
ググるとwindowsに以前からある問題らしい
ここを参考に設定をした
Windows10の重い遅い対策 - Qiita
やったのはMsMpEng.exeを検疫対象から外して
WSLのdataが入るディレクトリを丸ごと除外した
書き込んだファイルをすべてチェックしようとするため
書き込みの多いコマンドを使うとチェックが続いて
結果CPUの使用率が上がる
Trinityでは大量にfastqファイルが生成されるらしく
それがいちいち検疫される
この設定の結果、windows defender antivirus serviceの
CPU使用率がやや下がった
常に使用率1番だったのが順位が入れ替わるようになって
10-20%台まで落ちてる
少しでも他にリソースが回せるようになったのは助かる
RNA-seqその2、データのダウンロードと変換
シークエンスのデータの入手方法には
- 公共のデータベースからシークエンスデータをダウンロードする
- シークエンスを行う
などがある。
今回は公共のデータベースのデータを使う
公共データベースによって登録されているファイル形式は異なる
NCBIはSRA、DDBJ/EMBLEはfastq形式である
シークエンスの結果は一般にはfastq形式で
SRAはFastqに情報を追加して圧縮した形式である
解析にはFastq形式を使用するので、ツールを用いてSRAからFastqに変換をする必要がある。
解析に使用するシークエンスデータが複数ある場合
ファイル名を記入したリストファイルの作成してあると一括で処理できる。
まずはダウンロード。
Home - SRA - NCBI
例題としてHeLaのRNAseqを探す
検索窓に"HeLa RNAseq"と入れて検索すると312個引っかかった
試しにこのデータを使ってみる
RNA-seq of HeLa cells - SRA - NCBI
しかし、データの評価の仕方がまだよくわからない
これを選んだのはなんとなく。。。
データのアクセッションナンバーはSRR6799791で、
アドレスはここで探せる
SRA Explorer
実際に探したアドレスを使ってファイルをダウンロード
$ cd $HOME/usr/data/ $ mkdir ./sra/ $ cd ./sra/ $ wget ftp://ftp-trace.ncbi.nlm.nih.gov/sra/sra-instant/reads/ByRun/sra/SRR/SRR679/SRR6799791/SRR6799791.sra
サイズが2.9 GBあるので時間がそれなりに掛かる
"$HOME"は、チルダで書くのがいいんだろうけど
スクリプト内で"$HOME"を使っているのでこの表記で。
SRA Toolkitを入れてればコマンドも使えるようだけど
使ったことがないので割愛
SRA Toolkit 使い方 公開データのダウンロードとsra fastq変換 – バイオインフォ 道場 [bioinfo-Dojo]
ダウンロードしたファイルはSRA Toolkitsに含まれる
fastq-dumpを使ってfastqに変換する
SRA Toolkitは前回インストール済み
RNA-seqその1、インストール - mecobalamin’s diary
16 April 2020 追記
SRA Explorerで生成したアドレスではダウンロードできなくなっているっぽい
SRA toolkitのprefetchでダウンロードする
prefetchを実行する前にvdb-confingを実行する
Toolkit Documentation : Software : Sequence Read Archive : NCBI/NLM/NIH
vdb-configのあるディレクトリで以下のように実行する
./vdb-config -i
RAMのサイズ以外は変更していない
追記ここまで
fastq-dumpで変換しただけでは動かないコマンドがあったりする
実際Trinityが動かなかった
NCBI SRA Toolkitの使い方 - アメリエフのブログ
Trinityはアセンブルを行うツール
詳しくはまた今度。
で、sraファイルを複数置換できるようにスクリプトを書いた
まずsraファイルのリストを作成
$ ls $HOME/usr/data/sra/ > $HOME/usr/scripts/list_sra.txt
以下のスクリプトこの名前"RNAseq01_SratoFastq.sh"で保存
保存場所は$HOME/usr/scripts/
#!/bin/bash echo "change directory to $HOME/usr/scripts/" cd $HOME/usr/scripts/ w_dir="$HOME/usr/data" scripts_dir="$HOME/usr/scripts" sra_dir="${w_dir}/sra" out_dir="${w_dir}/raw" list_name="list_sra.txt" list_dir="$HOME/usr/scripts" echo "making directory" mkdir -p ${out_dir} echo "run fastq-dump" for i in $(cat ${list_dir}/${list_name} | sed 's/\.sra//g') do file=${list_dir}/${i}.sra fastq-dump \ --split-files \ --defline-seq '@$sn[_$rn]/$ri' \ --outdir ${out_dir} \ ${sra_dir}/${i}.sra sed -i -e 's/_forward//' ${out_dir}/${i}_1.fastq sed -i -e 's/_reverse//' ${out_dir}/${i}_2.fastq done
"--defline-seq"でRead1とRead2の区別がつくように、
配列名の最後に/1または/2をつけている
NCBI SRA Toolkitの使い方 - アメリエフのブログ
元データがペアエンドのデータなので"--split-files"を指定する
シェルスクリプトの実行方法はこちら
$ cd $HOME/usr/scripts/ $ bash RNAseq01_SratoFastq.sh
ファイルに実行権限を与えて実行しても良い
$ cd $HOME/usr/scripts/ $ chmod 755 RNAseq01_SratoFastq.sh $ ./RNAseq01_SratoFastq.sh
chmodの実行は最初の一回だけ行う
【 chmod 】コマンド――ファイル/ディレクトリのパーミッション(許可属性)を変更する:Linux基本コマンドTips(14) - @IT
結果はrawに保存される
ファイル名はSRR6799791_1.fastq及びSRR6799791_2.fastqとなる
1、2には対になるリードがそれぞれ保存されている
十分にメモリとストレージがあれば、このままでいいのだが、
使っている環境に対してファイルサイズが大きすぎるのでファイルを分割する
fastq形式は1つのリードを4行で表示するので
4行単位で分割する
ここを参考にしてスクリプトを書いた
大きいfastqファイルを分割マッピング(tophat, linux) - script of bioinformatics
fastqを分割するツールもあった
fastq / fastaの操作ツール seqkit - macでインフォマティクス
とりあえずファイルを10分割する
ファイル名は"RNAseq02_divideFastqFiles.sh"
このスクリプトを実行すると10個に分割されたfastqと
ファイルリストのファイル"list_div_SRR6799791"が作成される
#!/bin/bash echo "change directory to $HOME/usr/data/raw/" cd $HOME/usr/data/raw/ scripts_dir="$HOME/usr/scripts" w_dir="$HOME/usr/data" out_dir="${w_dir}/raw" list_name="list_sra.txt" list_dir="$HOME/usr/scripts" n=10 for i in $(cat ${scripts_dir}/list_sra.txt | sed 's/\.sra//g') do div_file="div_${i}" echo "count row number" read=$(grep "\+${i}" $HOME/usr/data/raw/${i}_1.fastq | wc -l) sp=$(expr \( \( ${read} \/ ${n} \) + 1 \) \* 4) echo "making directory" mkdir -p $HOME/usr/data/raw/${div_file}/ echo "read=${read}, sp=${sp}" echo "dividing fastq file" for j in {1..2} do split --numeric-suffixes=01 -l ${sp} $HOME/usr/data/raw/${i}_${j}.fastq ${div_file}_${j}_ mv ${div_file}_${j}_* ./${div_file} done ls ${out_dir}/${div_file}/ > ${out_dir}/${div_file}/list_${div_file}.txt done
RNA-seqその1、インストール
wsl上でRNA-seqの解析を行う
手順を教えてもらったのでそのメモ
メモするのは
- コマンドのインストール(このページ)
- RNA-seqその2、データのダウンロードと変換 - mecobalamin’s diary
- RNA-seqその3、trimmomatic - mecobalamin’s diary
- RNA-seqその4、Fastqファイルのマージ - mecobalamin’s diary
- RNA-seqその5、Hisat2でマッピング - mecobalamin’s diary
- RNA-seqその6、featureCountsでリードカウント - mecobalamin’s diary
- RNA-seqその7、TrinityでアセンブルしてBlastにかける - mecobalamin’s diary
- RNA-seqその8、edgeRを使ったTMM正規化 - mecobalamin’s diary
- RNA-seqその9、DESeq2を使った正規化 - mecobalamin’s diary
- RNA-seqその10、Dockerで環境構築 - mecobalamin’s diary
教えてもらったのは1-7まで。
8と9はウェブから情報を集めて自分でやってみた。
それと表示に必要なJBrowseのインストールなど
- JBrowseのインストール、三度目の正直 - mecobalamin’s diary
- JBrowseでbamファイルを表示する - mecobalamin’s diary
- bigwigの作成と表示 - mecobalamin’s diary
- JBrowseにplug-inを追加する - mecobalamin’s diary
- hg38のfastaとgtf/gff3 - mecobalamin’s diary
ファイルの整形だったりクオリティのチェックだったり
他にもやっているけど大まかにはこんなところ
目的は実行に使うコマンドが動くコードを記録すること
教えてもらった元のコードはクラスターコンピュータで動いていた
そのコードをノートPCのwslで動くように書き換えた
コードの検証のため計算結果とグラフを載せるが、
結果の検証は実力不足でできてない(特に自分でやってみた正規化のあたり)
コードが動く ≠ 計算結果が正しい、ということに注意したい
Dockerで環境を統一できたら
結果の検証に集中できると思う(けどやってない)
ここではwslでの実行に必要なコマンドのインストールと設定を記載する
このあたりのサイトも参考にした
RNA-Seq | 遺伝子発現量解析
使用しているpcのスペック
CPU: Intel Core i5-7200U CPU @ 2.5 GHz
Memory: 24.0 GB (8 GB + 16 GB)
Storage: 250 GB SSD + 500 GB HDD -> 960 GB SDD + 1 T SSD
OS: Windows 10 Home version 1803 build 17134.523
WSL: 18.04.1 LTS
メモリを増設してあるがいたって普通のノートPC、
と思っているけどそうでもないらしい
【山田祥平のRe:config.sys】普通のPCが普通に買えない - PC Watch
CPUはこのスペックでも1日とかで終わってくれる
メモリとストレージは多いほどいいけどノートPCだとちょっと厳しいか
SSDも安くなってきてるからいずれ交換できればいいが。
それとwslはファイルのやり取りがものすごく遅いらしいので
HDDよりはSDD、外付けよりも内蔵のストレージを使いたいが問題がある
いくつかのコマンドはファイルの読み書きがとても多く
タスクマネージャーを見てるとSSDの寿命に影響しそう
1日中ディスクにアクセスとかやってくれる
なのでRAMディスク、外付けストレージをうまく使う
あとwslを使うときの注意点がある
ファイルシステムの関係でwindows側からwslのファイルを編集してはいけないらしい
実際ファイルがおかしくなったことがあった
今後のアップデートで変わる可能性があるらしいけど、現状は不可っぽい
出力されたファイルや設定ファイルなどをwindowsのエディタ等から編集しない
編集する場合はファイルをコピーして行う
次にコマンド・プログラムのインストールについて。
wslでインストールするコマンドと
windowsにインストールするプログラムとがある
wslにインストールするのは
windowsにインストールするのは
- R
- Rのライブラリ
まずwslで使用するコマンドのインストールする
アーカイブのダウンロードして、解凍、リンクを貼る、
ということをコマンド毎にする
コマンドのダウンロードはwgetを使う
【 wget 】コマンド――URLを指定してファイルをダウンロードする:Linux基本コマンドTips(24) - @IT
ダウンローダーは他にもあるようですが。。。
解凍は圧縮形式に合わせる
【 tar 】コマンド――アーカイブファイルを作成する/展開する:Linux基本コマンドTips(40) - @IT
【 unzip 】コマンド――ZIPファイルからファイルを取り出す:Linux基本コマンドTips(35) - @IT
リンクは$HOME/usr/localに展開したコマンドファイルから
パスの通っている$HOME/binにシンボリックリンクを貼っている
細かいルールを知らないので自己流かもだけど。。。
ちなみにwslでパスは$HOME/.bashrcに記入する
export PATH="$HOME/bin:$HOME/miniconda3/bin:$PATH"
ファイルがなければ作る
bashでパスを書く設定ファイルの選び方にも
なんかルールがあるっぽいがとりあえず。
パスは先に書かれている方から優先順位が高い
実際にコマンドをインストールする
- sra toolkit
NCBIにアップロードされている
シークエンスのデータはsra形式になっている
これを展開するにはsra toolkitを使う
ダウンロード先と参考にしたサイトはこちら
https://www.ncbi.nlm.nih.gov/sra/docs/toolkitsoft/
NCBI SRA Toolkitの使い方 - アメリエフのブログ
$ wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/current/sratoolkit.current-ubuntu64.tar.gz
$ tar -zxvf sratoolkit.current-ubuntu64.tar.gz
シンボリックリンクを貼る
$ cp sratoolkit.2.9.2-ubuntu64/ ~/usr/local/ $ cd ~/bin $ ln -fs $HOME/usr/local/sratoolkit.2.9.2-ubuntu64/bin/fastq-dump ./
- prinseq-lite, FastQC, featureCount
prinseq-liteとFastQCもインストールしてリンクを貼る
prinseq-lite
PRINSEQ - Browse /standalone at SourceForge.net
PRINSEQ @ SourceForge.net
[Download] -> [Standalone] -> [prinseq-lite-0.20.4.tar.gz]をダウンロードする
prinseq-lite.plのパーミッションを変更する
FastQC
Babraham Bioinformatics - FastQC A Quality Control tool for High Throughput Sequence Data
[Download Now] -> [FastQC v0.11.8 (Win/Linux zip file)]をダウンロードする
展開したファイルのfastqcのパーミッションを変更する
$ chmod 755 fastqc
featureCountも同様にインストールする
mecobalamin.hatenablog.com
- miniconda
次にminicondaを使ってpythonコマンドをインストールする
trimmomaticとtrinityはbiocondaのパッケージのひとつで、
minicondaをまずインストールして必要なパッケージを追加する
anacondaとminicondaの比較
https://echomist.com/anaconda-vs-miniconda/
minicondaは3.x系をインストールする
RNA-seqで使うコマンドによっては2.x系が必要な場合もあるが、
その時は2.x系の環境をつくってインストールする
念の為minicondaのpython3から$HOME/binにリンクを張って3.x系を使うようにしておく
$ ln -s $HOME/miniconda/bin/python $HOME/bin
minicondaの配布元とインストール用シェルスクリプトのアドレス
Miniconda — Conda documentation
https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh
シェルスクリプトをダウンロードして実行する
$ wget https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh $ bash Miniconda3-latest-Linux-x86_64.sh
実行後はターミナルを再起動してパスを読み込ませる
.bashrcにパスが書かれている場合はを.bashrcを再読込する
.bashrcの再読込のコマンドはこれ
$ source $HOME/.bashrc
- anaconda package, Trinity、fastq-stats、numpy、hisat2及びstringtie
パッケージはここからインストール元を探す
https://anaconda.org/
パッケージ名で検索をかけてダウンロード方法を探す
例えばtrimmomaticならインストール元はここで
Trimmomatic :: Anaconda Cloud
インストール方法を見るとこう書かれてる
$ conda install -c bioconda trimmomatic
biocondaはchannel名で、trimmomaticがpackage名で、
channelが登録されているかを確認するには
$ conda config --get channel
登録されてなければ次のようにして登録する
$ conda config --add channel <channel名>
チャンネルによってインストールされる内容が違っていたりする
一緒にインストールされるパッケージのバージョンが変わったり
インストールされたりされなかったりもある
必要なパッケージが揃っているかの確認も必要
追加するのはTrinity、ea-utils、fastq-stats、numpy、hisat2及びstringtie
$ conda install -c bioconda trinity $ conda install -c bioconda ea-utils $ conda install -c bioconda numpy $ conda install -c bioconda hisat2 $ conda install -c bioconda stringtie
- salmon
trinityでsalmonを使うが、biocondaから配布されているのは
バージョンが古い(v0.8.1)のでオリジナルの配布元から
binaryをダウロードして使う(v.0.12.0)
Overview – Salmon: Fast, accurate and bias-aware transcript quantification from RNA-seq data
$ wget https://github.com/COMBINE-lab/salmon/releases/download/v0.12.0/salmon-0.12.0_linux_x86_64.tar.gz
miniconda3/binにリンクを貼る
$ cp salmon-0.12.0_linux_x86_64/ $HOME/usr/local/ $ ln -s $HOME/usr/local/salmon-0.12.0_linux_x86_64/bin/salmon $HOME/miniconda3/bin/
先にv0.8.1のリンクがbinに貼られている場合はlnコマンドの前にリンクを解除する
$ unlink $HOME/miniconda/bin/salmon
29 May 2019追記
hisat2とstringtieを上記のコマンドでインストールすると
pythonのバージョンを2.7にしていたようだ
確認不足だった
この2つのコマンドはバイナリーを使ったほうがいいかも
hisat2
https://ccb.jhu.edu/software/hisat2/index.shtml
バイナリーのリンク
http://ccb.jhu.edu/software/hisat2/dl/hisat2-2.1.0-Linux_x86_64.zip
stringtie
StringTie
バイナリーのリンク
http://ccb.jhu.edu/software/stringtie/dl/stringtie-1.3.6.Linux_x86_64.tar.gz
それぞれwgetでダウンロードしてファイルを展開、
$HOME/binにシンボリックリンクを貼る
condaでインストールして使う場合はpython3とpython2の環境を分ける
別に記事を書く
mecobalamin.hatenablog.com
追記ここまで
23 April 2020 追記
Hisat2とstringtieをインストールしたら
python3にインストールされた
もしかしたら切り替えが必要なくなったのかも
追記ここまで
これで全部、のはず。。。
足りないのは適宜インストールする
インストールしたはずのパッケージが見つからない時がある
condaは3.xでそっちにインストールしているのに
wslに元から入っているpythonが2.xだったりして
パスの関係でwslのpythonで実行されていると見つからないってなる
以前そんな事があって、そのときはパスの優先順位を変えた
mecobalamin.hatenablog.com
pipを使う方法もあるが、anacondaと併用してはいけないそうだ
注意することかも
condaとpip:混ぜるな危険 - onoz000’s blog
次にwindowsで動かすプログラム
- R
Windows版をインストールする
The Comprehensive R Archive Network
RGuiか、AtomでR scriptでRのスクリプトを実行できるようにする
- edgeR
Rに必要なライブラリをインストールする
bioconductorを利用してインストールする
mecobalamin.hatenablog.com
- Atom (必要なら)
エディタで、パッケージの追加でRコマンドをスクリプトを書きつつ実行できる
atom.io
学生の頃はEmacsだったけど宗旨替え
wslではvi
/path/to/R/R-3.x.x/Rscript.exeにwindowsの環境変数でパスを通す
Rのアップデートをするたびに通し直す
mecobalamin.hatenablog.com
- script
- language-r
scriptでRを実行するのに必要
ATOMをR言語に対応させる方法 - Qiita
パッケージの更新が遅れているようで
設定ファイルにエラーが残ってるがそのままでも問題ない
修正方法もあったので直してあるけどそのサイトが見つからない。。。
JBrowseのインストール 続き
24 February 2019 追記
やり直したので新しい方のリンク
mecobalamin.hatenablog.com
xlocale.hがなくてJBrowseのインストールで詰まった
前回も参照したサイトに方法があった。
xlocale.h not found on Ubuntu while installing · Issue #16 · agracio/electron-edge-js · GitHub
無理やりだけどリンクを貼る。
$ sudo ln -s /usr/include/locale.h /usr/include/xlocale.h
今度はdb.hがない
まとめてエラー出してくれよ―
あってるかわからないけどminicondaのlibdbに同じ名前のがあったので
これもリンクを貼る
$ sudo ln -s /home/hogehoge/miniconda3/pkgs/libdb-6.1.26-0/include/db.h /usr/include/
condaでlibdbのパッケージをインストールしても良さそう
$ conda install -c bioconda libdb
と、ここまで書いてみたのだが、
実際いろいろやってて忘れてきてるので
ざっとまとめてみる
手順は
- JBrowseのアーカイブを展開し、/var/www/html/以下などに移動する
- JBrowseのディレクトリのオーナーを変更する
- 必要なpluginを/jbrowse/pluginにコピーする
- package.jsonとyarn.lockを別ディレクトリにコピーをとっておく
- node.jsをインストールする
- /jbrowse/setup.shを実行する
- volvoxとyeastのデータディレクトリができれば成功
- データの入ったディレクトリを/jbrowse/にコピーする
- ウェブサーバー経由でブラウザに表示する
JBrowseの配布元はここ
jbrowse.org
setup.shを動かす前までの作業も紹介されてる
こんな感じ。
curl -O https://github.com/GMOD/jbrowse/releases/download/1.16.1-release/JBrowse-1.16.1.zip unzip JBrowse-1.16.1.zip sudo mv JBrowse-1.16.1 /var/www/html/jbrowse cd /var/www/html sudo chown `whoami` jbrowse cd jbrowse ./setup.sh # don't do sudo ./setup.sh
JBrowseのアーカイブにはpackage.jsonが含まれていて、
setup.shを実行するとpackage.jsonをもとに
必要なパッケージをインストールしてくれる
node.jsはここ
nodejs.org
npmも一緒にインストールされる
忘れずにパスを通す
前回も書いたけど、使うperlでエラーが出たりした。
Perlはubuntuにはじめから入っている(/usr/bin/)のが問題なさそう。
setup.shがエラーを吐かなかった。
minicondaのはコンパイラを示すディレクトリがなんか変で
コンパイルエラーが出る
インストールに問題があってやり直すときの注意点。
setup.shを何度も実行するとインストールされる
パッケージの依存関係がおかしくなる様子。
追記があるのかpackage.jsonとyarn.lockのサイズが変わってく。
なので、setup.shを実行する前に
package.jsonとyarn.lockを削除して、
アーカイブ展開直後の変更の加えられていない
ファイルと置き換えるとうまくいった
それと、setup.shの実行にsudoを使ってはいけないらしい
まずJBrowseのディレクトリ以下についてオーナーを変更する
それからsetup.shを実行する
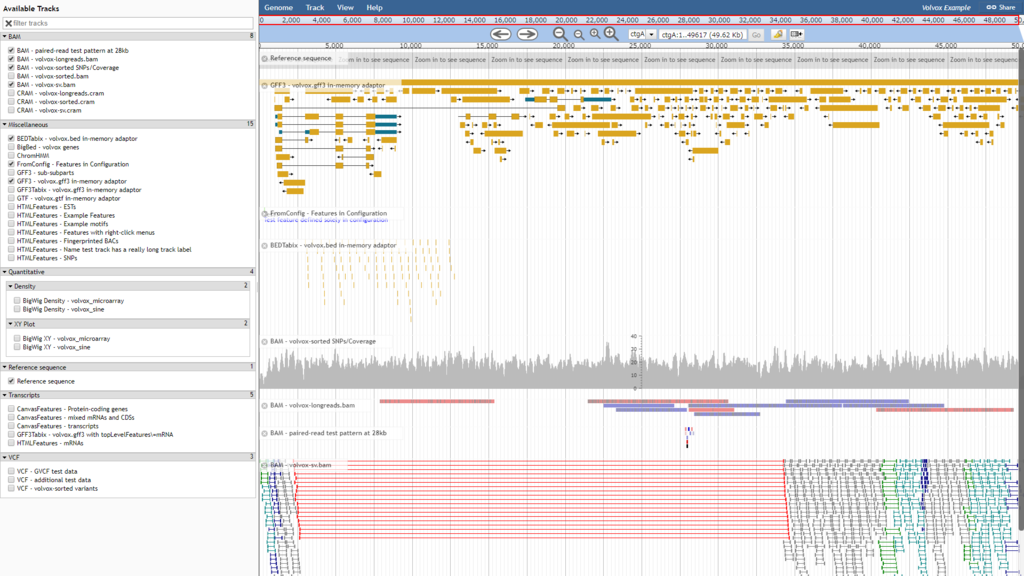
つくられたvolvoxとyeastのデータはブラウザで表示できる
以前やったみたいにpowershellとpythonを使って
ウェブサーバーを立ち上げる
mecobalamin.hatenablog.com
サーバー経由でファイルを読み込ませるとうまくいった
http://192.168.100.110:8080/jbrowse-1.16.1-release/index.html?data=sample_data%2Fjson%2Fvolvox
こんな感じに表示される
JBrowseのインストール
24 February 2019 追記
やり直したので新しい方のリンク
mecobalamin.hatenablog.com
ゲノムデータをブラウザで表示するツールにJBrowseがある。
JBrowse · A fast, embeddable genome browser built with HTML5 and JavaScript
元論文はこちら
JBrowse · A fast, embeddable genome browser built with HTML5 and JavaScript
仕事で必要なので、試しにwslにインストールした。
JBrowseのアーカイブをダウンロードして展開。
$ wget https://github.com/GMOD/jbrowse/archive/1.16.1-release.tar.gz
$ tar -zxvf 1.16.1-release.tar.gz
で、できたディレクトリjbrowse-1.16.1にある
setup.shを実行するだけで、セットアップ終了、
のはずなのだが、それ以外のところでいろいろ引っかかった。
このシェルスクリプトを動かすにはperlが必要。
別件でインストール済みだったminicondaの
perlをそのまま使うことにした。
結局perl入れなおすことになるのだが。
ちなみにminicondaのインストール方法は配布元から
シェルスクリプトを落としてきて実行するだけ。
配布元はこちら
Miniconda — Conda documentation
$ cd ~/ $ cd mkdir ./tmp $ cd ./tmp $ wget https://repo.continuum.io/miniconda/Miniconda3-latest-Linux-x86_64.sh $ bash Miniconda3-latest-Linux-x86_64.sh
perlも含まれている。
これでオッケーのはずだったのだが、
perl moduleのコンパイルエラーが出る。
logファイルにはこんな感じで出力されてる。
/bin/sh: 1: /tmp/build/80754af9/perl_1527832170752/_build_env/bin/x86_64-conda_cos6-linux-gnu-gcc: not found
x86_64-conda_cos6-linux-gnu-gccが見つからない。
condaでインストールできるらしい
x86_64-conda_cos6-linux-gnu-c++ command not found · Issue #770 · RcppCore/Rcpp · GitHub
minicondaにパッケージをインストールするときは、
ここから探す
:: Anaconda Cloud
$ conda install -c conda-forge/label/gcc7 gxx_linux-64
jbrowse-1.16.1/setup.shを実行するが、
まだコンパイルエラーが出る。。。
インストールされてないのはこれら。
! Installing the dependencies failed: Module 'Devel::Size' is not installed, Module 'DB_File' is not installed, Module 'Bio::FeatureIO' is not installed, Module 'DBD::SQLite' is not installed, Module 'Bio::SeqFeature::Annotated' is not installed, Module 'Heap::Simple::XS' is not installed, Module 'JSON::XS' is not installed, Module 'PerlIO::gzip' is not installed, Module 'DBI' is not installed
perl moduleのコンパイルエラーなので、
cpanを使ったインストールを試したけどそれもだめ。
ちなみにcpanとは
CPAN - Wikipedia
cpanのインストール方法は
$ perl -MCPAN -e shell
perl moduleのインストールは
$ cpan cpan[1]> install PerlIO::gzip
みたいにする
それでも同じようにコンパイルエラーが出る
上でも出したけどよくよく見るとコンパイラのpathがおかしい
こんなディレクトリないな
/tmp/build/80754af9/perl_1527832170752/_build_env/
~/miniconda3/lib/5.26.2/x86_64-linux-thread-multi/Config.pm
で指定されてるけどなんかいろいろ変だ。
別のPCに入れてあったminicondaを確認すると同じように
変なpathだったので、書き換えてしまったわけではなさそう。
Perlの入れ直しをする
$ conda install -c conda-forge perl
Config.pmで指定されているpathは"/usr/local/lib"になった
これでsetup.shを実行するとコンパイラは見つかったっぽいが。。。
今度はincludeファイルがないときた。。。
perl.h:738:13: fatal error: xlocale.h: そのようなファイルやディレクトリはありません # include <xlocale.h>
配布がなくなったみたい
xlocale.h not found on Ubuntu while installing · Issue #16 · agracio/electron-edge-js · GitHub
さてどうしたものか。。。
続きます
sedの使い方
ファイルサイズが10 MBで中身が10万行を
超えるようなテキストファイルで、
列のラベルによって置換する文字を
変えるシェルスクリプトを書いた
例えばn列がhogehogeの場合、m列をaaaからbbbに変えて
n列がhugahugaの場合、m列をaaaからcccに変えるような
シェルスクリプト
まずはgrepで置換する行を選んで、
sedで置換する方法でやった
一行ずつ結果を出力を書き込むので
時間がかかる。。。。
#!/bin/bash dir=`pwd` filename=largetext.txt filein=${dir}/${filename} fileout=${dir}/rename_${filename} filetmp=${dir}/tmp_${filename} :> $fileout while read line; do if echo $line | grep hogehoge > ./null; then echo $line | sed 's/aaa/bbb/' >> $fileout else echo $line >> $fileout fi done < $filein mv $fileout $filetmp :> $fileout while read line; do if echo $line | grep hugahuga > ./null; then echo $line | sed 's/aaa/ccc/' >> $fileout else echo $line >> $fileout fi done < $filetmp
人に聞いたらこのサイトを教えてもらった
sedでこういう時はどう書く? - Qiita
sedで特定の文字列を含む場合の条件付ができる
sed -e '/[パターン]/s/[置換前]/[置換後]/'
パターンにマッチする場合に
置換前の文字列を置換後の文字列に置換する
オプションを-rに変えると拡張正規表現で
パターンマッチができる
書き直したスクリプトがこれ
#!/bin/bash dir=`pwd` filename=largetext.txt filein=${dir}/${filename} fileout=${dir}/rename_${filename} filetmp=${dir}/tmp_${filename} cat ${filein} | sed '/hogehoge/s/aaa/bbb/' > ${fileout} mv ${fileout} ${filetmp} :> ${fileout} cat ${filetmp} | sed -e '/hugahuga/s/aaa/ccc/' > ${fileout} :> ${filetmp}
実行結果が分のオーダーから秒のオーダーまで短縮
スクリプトもスッキリしたし知らないって不幸
catの結果をsedで読み込んでいるけど
sedは引数にファイルを取れるので
もうちょっときれいに書けるかも
あと":"は何もしないってコマンドらしい
:> filename
で空のファイルを出力する